Datenvorbereitung
R für empirische Wissenschaften v1.0.2
Jan Philipp Nolte
Source:vignettes/02_Datenvorbereitung.Rmd
02_Datenvorbereitung.RmdEinführung
Die Methoden zur Datenvorbereitung zu lernen, ist der mit Abstand wichtigste und schwierigste Schritt auf dem Weg, R in der Praxis anwenden zu können. Deswegen ist dieser Teil auch der umfangreichste und mit einer interaktiven Übung am Ende versehen, an der Du Dein Wissen festigen kannst. Allerdings liegt in der Datenvorbereitung auch die größte Stärke im Vergleich zu Programmen wie SPSS. Bevor wir mit der Datenvorbereitung beginnen, müssen wir uns jedoch noch verschiedene Themen aneignen.
Projektorientierter Workflow
Projekte sind ein modernes Konzept, um R Scripte und Datensätze zu organisieren. Es gibt verschiedene Vorteile durch Projekte. Man kann zwischen Projekten innerhalb von R oben rechts einfach wechseln, der Ort von Datensätzen muss nicht mehr in Abhängigkeit vom eigenen Computer angepasst werden und Projekte sind aufgrund von in sich abgeschlossenen Umgebungen replizierbar. Aber wie erstellt man ein Projekt?
Oben rechts befindet sich ein Reiter namens
Project: (None). Klicke auf den Reiter und klicke aufNew Project.... In dem Reiter kann man auch schon bestehende Projekte sehen. Bei Klick auf die Projekte wird das Projekt sofort mit allen zuvor geöffneten R Scripten geöffnet. Ganz ohne suchen des Ordners auf Deinem Computer.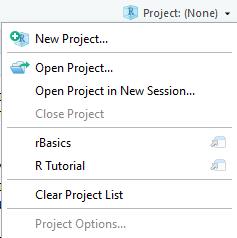
Nun sehen wir drei Möglichkeiten. Wir entscheiden uns exemplarisch für
New Directory, was einen neuen Ordner mit einer Projektdatei erstellen wird.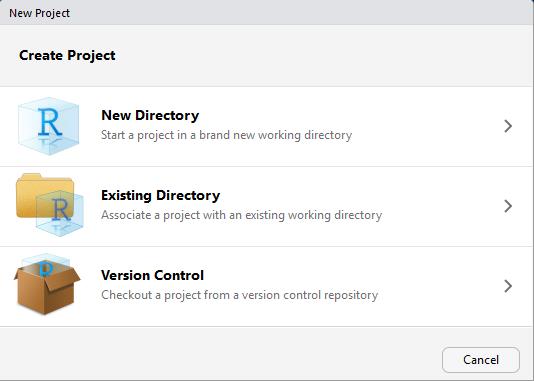
Bevor die Projektdatei erstellt wird, müsenn wir noch spezifizieren, welchen Projektyp wir haben möchten. Hier wählen wir
New Project.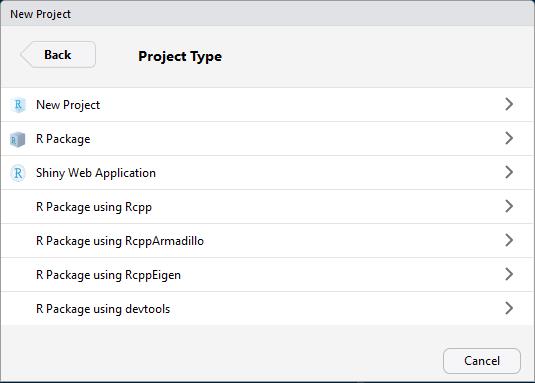
Nun müssen wir noch den Namen des neuen Ordners festlegen und angeben, wo auf dem Computer der Ordner erstellt werden soll. In Zukunft kann der Ordner nach belieben verschoben werden. Das wichtige ist die nun in diesem Ordner enthaltene Projektdatei (
projektname.rproj).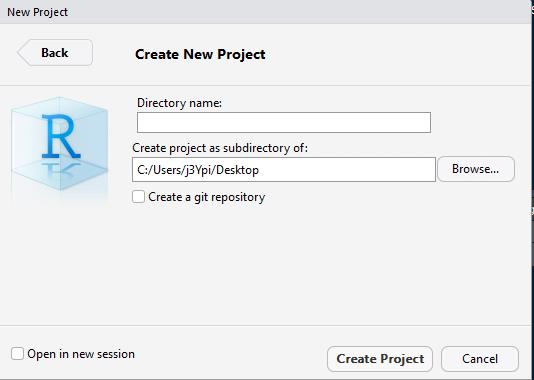
Wenn Du nun das Package here lädst, findet das Package
sofort den relativen Pfad zu Deiner Projektdatei heraus. Was bedeutet
das? Egal wo sich der Ordner befindet (Desktop, Downloads Ordner oder
tief verschachtel in Ordnerstrukturen), here findet den
richtigen Weg. Zu wissen, wo sich das benutze R Script auf dem Computer
befindet, ist kriegsentscheidend zum Einlesen des Datensatzes. Durch die
Projektdatei braucht man also nie wieder das Working Directory per Hand
mit setwd() festzulegen.
Der erste Schritt ist also immer das Erstellen eines R Projekts und das Laden des Packages am Anfang jedes neuen Scripts, mit dem man einen Datensatz einlesen möchte.
Aber wie kann man den Datensatz nun einlesen?
Import/Export
Auch wenn in diesem Tutorial die Datensätze bereits im Package enthalten sind, muss man in den meisten Fällen den Datensatz erst einmal einlesen. Datensätze können in verschiedenen Formaten vorliegen. Dies ist vor allem abhängig davon, mit welchen Programmen Unternehmen, Universitäten oder Kollegen arbeiten. Einige Beispiele sind:
- R (
.RData|.rda|.rds) - Excel (
.xlsx|.xls) - SPSS (
.sav) - Stata (
.dta) - Comma seperated values (
.csv) - Tab seperated values (
.tsv)
Einlesen eines Datensatzes
Eingelesen werden können sämtliche Dateientypen mithilfe von
import() aus dem rio Package. Dabei erkennt
die Funktion die Dateiendung und übernimmt hinter den Kulissen alles
weitere. Damit der Datensatz als tibble (siehe Kapitel
Extras) eingelesen wird, muss man
setclass = "tbl" setzen. Für den Moment musst Du Dir beim
setclass Argument noch nichts denken (aber trotzdem
verwenden!).
Dabei muss sich das test.xlsx Excel Sheet innerhalb des
selben Ordners wie die Projektdatei befinden. Der zweite Schritt ist
also immer rio zu laden, da sonst die Funktionen nicht
aufgerufen werden können.
Einlesen multipler Datensätze
Nicht selten kommt es vor, dass man nicht nur einen, sondern gleich
mehrere Datensätze einlesen möchte. Dabei werden die verschiedenen Files
in einer Liste (mehr zu Listen im Kapitel Extras)
gespeichert. dir() (für directory) listet alle Files
innerhalb eines Ordners auf. Der Pfeil
wird später erklärt.
files <- dir(pattern = "csv$")Das pattern Argument ist optional. Man kann damit zum
Beispiel die Dateiendung spezifizieren. Das Dollar-Zeichen am Ende
stellt sicher, dass csv am Ende des Dateinamen steht. Zum
Importieren verwenden wir hier import_list().
import_list(files, setclass = "tbl")Würden die Dateien in einem Unterordner data liegen,
könnte man mit der Funktion here() aus dem gleichnamigen
Package den Pfad dahin spezifizieren.
Sind die Datensätze im selben Format, kann man
import_list() das Argument rbind geben, sodass
alle Datensätze zeilenweise zusammengebunden werden.
import_list(here("data", files), setclass = "tbl", rbind = TRUE)import_list() funktioniert übrigens auch auch bei Excel
Workbooks, die mehrere Excel Sheets beinhalten. Dabei wird jedes Sheet
in einem Listenelement gespeichert. Für den Moment sind Listen für uns
allerdings nicht weiter von Bedeutung.
Speichern
Auch das Speichern von Datensätzen könnte in R durch das
rio Package intuitiver nicht sein. Anstelle von
import() muss man lediglich export() benutzen.
Das erste Argument der Funktion ist der Datensatzname, mit dem man
innerhalb von R auf selbigen zugreift. Das zweite der gewünschte
Dateienname.
export(datensatz, "test.xlsx")Zuweisungspfeil
Damit man den Datensatz auch speichert, muss man ihn einer Variable zuweisen. Dies macht man in R mit einem Zuweisungspfeil. Dabei wird die Variable in die lokale Environment gespeichert. Wichtig zu verstehen ist, dass in R nichts gespeichert wird, wenn man es nicht einer Variable zuweist.
Variablen kann man grundsätzlich fast so benennen wie man möchte. Man
darf nur nicht mit einer Zahl anfangen oder nach einem Punkt direkt eine
Zahl als Namen wählen wie bei .2VariablenName. Auf Umlaute
sollte im Zusammenhang mit Programmiersprachen ebenfalls
immer verzichtet werden. In diesem Fall ist der
Variablenname tipp_wm.
tipp_wm <- import("tipp_wm.xlsx", setclass = "tbl")Auch bei anderen Befehlen, die wir später kennenlernen, ist der Zuweisungspfeil zentral, um Ergebnisse zu speichern und sie später wieder zu verwenden.
Der tidyverse Ansatz
Das tidyverse ist eine Zusammenstellung mehrerer
Packages, die aufeinander abgestimmt sind. Gegenüber Base R
macht es vieles im Kontext von Datenverarbeitung erheblich einfacher.
Der Großteil der Aufgaben, die wir mithilfe von tidyverse
Packages machen werden, kann man auch mit normalen Base R
Funktionen ausführen. Allerdings kommt man mithilfe des
tidyverse einfacher und schneller ans Ziel, mit dem
Nebeneffekt einer nachvollziehbareren Syntax. Gerade wenn man sich
Monate später den eigenen Code noch einmal ansieht, lernt man den Wert
von Lesbarkeit zu schätzen. Zur Lesbarkeit trägt auch die angemessene
Verwendung von Leerzeichen bei.
Der Hintergrund vom tidyverse ist es, tidy
data zu kreieren. Man spricht von tidy data,
wenn:
- Jede Variable eine Spalte ist
- Jede Beobachtung eine Zeile ist
- Jeder Wert in einer Zelle ist
Wir halten fest: Nach dem Starten von RStudio erstellen wir ein
Projekt und laden erst einmal die drei wichtigsten Packages
here, rio und tidyverse.
Datentypen
Es gibt verschiedene Datentypen, die in den Spalten gespeichert werden. Die drei Hauptdatentypen, sind hier aufgelistet. In R werden sie etwas untypisch im Vergleich zu anderen Programmiersprachen benannt. In Klammern steht die jeweilige Abkürzung.
-
numeric(num / int / dbl): z.B. Alter, Gehalt
42## [1] 42-
character(chr): z.B. Herkunftsland, Allergien
"Hallo Welt"## [1] "Hallo Welt"-
logical(lgl): TRUE, FALSE
2 == 2## [1] TRUE
1 < 2 & 1 == 2## [1] FALSE
1 < 2 | 1 == 2## [1] TRUELogical Datentypen sind sicherlich erst einmal etwas seltsam für
Programmiereinsteiger. Grundsätzlich kann eine logische Aussage immer
nur TRUE oder FALSE zurückgeben. Ist ein Apfel
gleich einer Banane? FALSE! Ist 1 kleiner als 2?
TRUE! Bei logischen Abfragen ist es wichtig zu beachten,
dass, wenn man auf Gleichheit von zwei Werten prüft, nicht ein einfaches
=, sondern ein doppeltes == nötig ist. Man
kann logische Aussagen auch kombinieren. Wenn beide logische Abfragen
stimmen sollen, kann man ein & (UND) schreiben. Wenn
nur einer Aussage richtig sein muss, damit der Wert TRUE
zurückgegeben wird, muss man | (ODER) schreiben.
Ungleichheit kann man mit != abfragen. Das Konzept
logischer Datentypen ist sehr wichtig für uns, also merken und damit
warm werden!
Datenvorbereitung
Wir erinnern uns, bei der Datenvorbereitung immer das
tidyverse zu laden. Schauen wir uns zuerst einmal den
ersten Datensatz an. Wer hat sich bei der Fußball Weltmeisterschafft
2018 in Russland nicht häufig über die Ergebnisse gewundert? Unseren 6
Personen in der Tipprunde in unserem Datensatz ging es nicht anders.
Drei der Urväter der zwei großen Strömungen der modernen Statistik haben sich aus dem Grabe erhoben, um noch einmal beim Tippen richtig abzuräumen. Reverant Thomas Bayes, Jerzy Neyman und Egon Pearson stellen sich dem Expertenwissen der drei Studenten Alice, Bob und Charlie.
Um den Datensatz ausgeben zu lassen, kannst Du einfach den Datensatznamen ausführen.
tipp_wm## # A tibble: 384 × 7
## Spieltag Person Tipp Ergebnis Tipp_Richtung Wahre_Richtung Art
## <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 1 Thomas_Bayes 2:0 5:0 S S Gruppenpha…
## 2 2 Thomas_Bayes 1:3 0:1 N N Gruppenpha…
## 3 2 Thomas_Bayes 0:0 0:1 U N Gruppenpha…
## 4 2 Thomas_Bayes 1:2 3:3 N U Gruppenpha…
## 5 3 Thomas_Bayes 2:1 2:1 S S Gruppenpha…
## 6 3 Thomas_Bayes 3:0 1:1 S U Gruppenpha…
## 7 3 Thomas_Bayes 0:2 0:1 N N Gruppenpha…
## 8 3 Thomas_Bayes 4:0 2:0 S S Gruppenpha…
## 9 4 Thomas_Bayes 2:1 0:1 S N Gruppenpha…
## 10 4 Thomas_Bayes 3:0 0:1 S N Gruppenpha…
## # … with 374 more rowsWir sehen sofort, es gibt 384 Zeilen und 7 Spalten. Es werden immer nur die ersten 10 Werte angezeigt.
- Spieltag: Nummer des Spieltags. An jedem Spieltag haben 1 bis 4 Spiele statt gefunden.
- Person: Die 6 verschiedenen Tipper
- Tipp: Der abgegebene Tipp für das jeweilige Spiel
- Ergebnis: Das tatsächliche Ergebnis
- Tipp_Richtung: Tipp, ob Sieg/Niederlage oder Unentschieden ausgehend von der Heimmannschaft
- Wahre_Richtung: Tatsächliche Richtung, ob Sieg/Niederlage oder Unentschieden ausgehend von der Heimmannschaft
- Art: Art des Spiels (Gruppenphase, Achtelfinale, etc.)
(Der Datensatz ist in der Form nicht tidy.)
Du wirst schnell sehen, wie sehr sich die Methoden des
tidyverse am normalen Englisch orientieren. Wenn wir zum
Beispiel nur auf die Tipps von Reverent Bayes schauen möchten, können
wir mit filter() einen einfach Filter setzen. Dabei müssen
wir nur die Spalte mit der Information über die Person auswählen und mit
einem doppelten Gleichheitszeichen abfragen, ob diese Herrn Bayes
entspricht.
## # A tibble: 64 × 7
## Spieltag Person Tipp Ergebnis Tipp_Richtung Wahre_Richtung Art
## <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 1 Thomas_Bayes 2:0 5:0 S S Gruppenpha…
## 2 2 Thomas_Bayes 1:3 0:1 N N Gruppenpha…
## 3 2 Thomas_Bayes 0:0 0:1 U N Gruppenpha…
## 4 2 Thomas_Bayes 1:2 3:3 N U Gruppenpha…
## 5 3 Thomas_Bayes 2:1 2:1 S S Gruppenpha…
## 6 3 Thomas_Bayes 3:0 1:1 S U Gruppenpha…
## 7 3 Thomas_Bayes 0:2 0:1 N N Gruppenpha…
## 8 3 Thomas_Bayes 4:0 2:0 S S Gruppenpha…
## 9 4 Thomas_Bayes 2:1 0:1 S N Gruppenpha…
## 10 4 Thomas_Bayes 3:0 0:1 S N Gruppenpha…
## # … with 54 more rowsWir sehen, dass nur noch 64 Spalten enthalten sind. So weit so gut,
aber was ist dieses kryptische “Prozent, größer als, Prozent” (
%>% )? Was erst einmal nach Hacken ala Mr. Robot
aussieht, bindet ganz einfach nur zwei Code Zeilen aneinander. Wir
werden gleich sehen, worin der große Vorteil davon besteht. Man
bezeichnet diese Zeichnenkombination als Pipe. Am
Anfang jeder Pipe steht immer der Name des Datensatz. In unserem Fall
tipp_wm. Ab hier weiß R für alle mit der Pipe verbunden
Funktionen, welcher Datensatz gemeint ist. Deswegen müssen wir
filter() auch nicht noch einmal extra mitteilen, in welchen
Datensatz nach der Spalte Person gesucht werden soll.
Um dieses Monster zu erschaffen, muss man allerdings nicht
immer % und > und % einzelnd eingeben sondern kann einfach
crtl + shift + M auf Windows oder
cmd + shift + M auf Mac schreiben. Shortcuts sind des
Programmierers bester Freund.
Schauen wir uns direkt das nächste Beispiel an. Wollen wir nun die
häufige Aufgabe ausführen, eine neue Spalte zu erstellen, können wir
dies mit mutate() machen. Auch hier kann man sich den
Befehl gut mit der deutschen Übersetzung des Wortes als etwas verändern,
merken. Wie immer steht der Datensatzname ganz oben noch vor der ersten
Pipe. Wie für alle kommenden Funktionen zur Datenmanipulation muss auch
hier der Datensatzname nicht extra innerhalb von mutate()
aufgerufen werden. Das geschieht alles hinter den Kulissen. In diesem
Beispiel nehmen wir willkürlich die Spieltag Spalte und
addieren Zeilenweise eine 1 hinzu (würde uns in der Praxis natürlich
nichts bringen) und nennen diese neue Spalte kreativer Weise
Neue_Spalte. Der Name vor dem Gleichheitszeichen ist immer
der Name der neuen Spalte.
## # A tibble: 384 × 8
## Spieltag Person Tipp Ergebnis Tipp_Richtung Wahre_Rich…¹ Art Neue_…²
## <dbl> <chr> <chr> <chr> <chr> <chr> <chr> <dbl>
## 1 1 Thomas_Bayes 2:0 5:0 S S Grup… 2
## 2 2 Thomas_Bayes 1:3 0:1 N N Grup… 3
## 3 2 Thomas_Bayes 0:0 0:1 U N Grup… 3
## 4 2 Thomas_Bayes 1:2 3:3 N U Grup… 3
## 5 3 Thomas_Bayes 2:1 2:1 S S Grup… 4
## 6 3 Thomas_Bayes 3:0 1:1 S U Grup… 4
## 7 3 Thomas_Bayes 0:2 0:1 N N Grup… 4
## 8 3 Thomas_Bayes 4:0 2:0 S S Grup… 4
## 9 4 Thomas_Bayes 2:1 0:1 S N Grup… 5
## 10 4 Thomas_Bayes 3:0 0:1 S N Grup… 5
## # … with 374 more rows, and abbreviated variable names ¹Wahre_Richtung,
## # ²Neue_SpalteMöchten wir nun bestimmte Spalten auswählen, können wir das einfach
mit select() machen. Erneut nach genau dem selben Schema.
Hier wählen wir die Spalten Spieltag, Person
und Ergebnis aus. Man kann an dieser Stelle auch Spalten
umbenennen, indem man den neuen Namen einfach vor den Spaltennamen
schreibt. Wir machen exemplarisch aus Person einfach mal
Tipper.
## # A tibble: 384 × 3
## Spieltag Tipper Ergebnis
## <dbl> <chr> <chr>
## 1 1 Thomas_Bayes 5:0
## 2 2 Thomas_Bayes 0:1
## 3 2 Thomas_Bayes 0:1
## 4 2 Thomas_Bayes 3:3
## 5 3 Thomas_Bayes 2:1
## 6 3 Thomas_Bayes 1:1
## 7 3 Thomas_Bayes 0:1
## 8 3 Thomas_Bayes 2:0
## 9 4 Thomas_Bayes 0:1
## 10 4 Thomas_Bayes 0:1
## # … with 374 more rowsManchmal möchte man aber Spalten umbennen und alle Spalten im
Datensatz behalten. Wir erinnern uns, select() wählt nur
die explizit genannten aus. Eine Lösung bietet rename() mit
genau der gleichen Syntax, nur werden hier alle weiteren Spalten
behalten.
## # A tibble: 384 × 7
## Spieltag Tipper Tipp Ergebnis Tipp_Richtung Wahre_Richtung Art
## <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 1 Thomas_Bayes 2:0 5:0 S S Gruppenpha…
## 2 2 Thomas_Bayes 1:3 0:1 N N Gruppenpha…
## 3 2 Thomas_Bayes 0:0 0:1 U N Gruppenpha…
## 4 2 Thomas_Bayes 1:2 3:3 N U Gruppenpha…
## 5 3 Thomas_Bayes 2:1 2:1 S S Gruppenpha…
## 6 3 Thomas_Bayes 3:0 1:1 S U Gruppenpha…
## 7 3 Thomas_Bayes 0:2 0:1 N N Gruppenpha…
## 8 3 Thomas_Bayes 4:0 2:0 S S Gruppenpha…
## 9 4 Thomas_Bayes 2:1 0:1 S N Gruppenpha…
## 10 4 Thomas_Bayes 3:0 0:1 S N Gruppenpha…
## # … with 374 more rowsSo weit so gut. Aber wozu brauchen wir nun diese kryptische Pipe? Es
ist äußerst selten, dass man nur einen Befehl auf einmal anwenden
möchte. Wenn wir zum Beispiel die drei gelernten Funktionen innerhalb
eines Befehls kombinieren möchten, können wir diese einfach mit einer
Pipe verbinden. Wir erstellen also zuerst eine neue Spalte, filtern alle
Einträge von Bob heraus und schauen uns dann nur die drei Spalten
Person, Ergebnis und Neue_Spalte
an.
tipp_wm %>%
mutate(Neue_Spalte = Spieltag + 1) %>%
filter(Person == "Bob") %>%
select(Person, Ergebnis, Neue_Spalte)## # A tibble: 64 × 3
## Person Ergebnis Neue_Spalte
## <chr> <chr> <dbl>
## 1 Bob 5:0 2
## 2 Bob 0:1 3
## 3 Bob 0:1 3
## 4 Bob 3:3 3
## 5 Bob 2:1 4
## 6 Bob 1:1 4
## 7 Bob 0:1 4
## 8 Bob 2:0 4
## 9 Bob 0:1 5
## 10 Bob 0:1 5
## # … with 54 more rowsDie Reihenfolge der Funktionsaufrufe spielt dabei eine entscheidene
Rolle. Hätten wir zuerst nur die Spalten
Ergebnis und Neue_Spalte ausgewählt, hätten
wir anschließend nicht mehr nach Bob suchen können, da die Spalte
Person gar nicht mehr im Datensatz enthalten wäre (siehe
das select() Beispiel oben). Außerdem wäre die
Neue_Spalte noch gar nicht existent.
Es gibt noch mehr nützliche Funktionen – alle nach dem selben
Prinzip. Möchten wir die Variablen gruppieren, um für jede der Gruppen
eine bestimmte Funktion anzuwenden, verwenden wir
group_by(). Beispielhaft zählen wir die Anzahl der Einträge
jeder Person mit count(). Um die Reihenfolge der Personen
in absteigender Reihenfolge des Alphabets anzuzeigen, können wir
innerhalb von arrange() die desc()
(descending, engl. absteigend) Funktion verwenden.
## # A tibble: 6 × 2
## # Groups: Person [6]
## Person n
## <chr> <int>
## 1 Thomas_Bayes 64
## 2 Jerzy_Neyman 64
## 3 Egon_Pearson 64
## 4 Charlie 64
## 5 Bob 64
## 6 Alice 64Hier hat jede Person gleich viele Tipps abgegeben. Zumindest befinden
sich gleich viele Zeilen für jede Person im Datensatz. Aufschluss über
die Anzahl der NAs kriegen wir erst später (siehe Kapitel
Deskriptive Statistik).
Bei ANOVAs braucht man Kategorien, nach denen man die abhängige
Variable untersuchen kann. Was wäre, wenn wir untersuchen möchten, ob
sich die erreichten Punktzahlen – also die Qualität der Tipps – zwischen
Gruppenphase und KO-Phase verändert haben? In der Spalte
Art gibt es allerdings die Ausprägungen
Gruppenphase, Achtelfinale,
Viertelfinale, Halbfinale und
Finale. Alle Finalarten fallen in die Kategorie KO-Phase.
Was wir brauchen, ist also eine neue Spalte, die immer
wenn Gruppenphase in der Spalte
Art steht, Gruppe beinhaltet und
ansonsten KO. Erreichen können wir dies
mit dem if_else() (engl. für wenn_ansonsten) Befehl. Als
erstes Argument muss der Funktion die Bedingung gegeben werden, die
erfüllt werden soll. Diese muss immer einen logischen Datentyp
zurückgeben – also wahr oder falsch (TRUE,
FALSE). Das zweite Argument ist das, was passieren soll,
wenn die Bedingungen zutrifft. Das dritte Argument
kümmert sich um alle sonstigen Fälle. Also
wenn in der Spalte Art die
Gruppenphase steht, trage Gruppe ein,
ansonsten KO. Wir nennen diese neue Spalte
Phase.
## # A tibble: 384 × 8
## Spieltag Person Tipp Ergebnis Tipp_Richtung Wahre_Richtung Art Phase
## <dbl> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 1 Thomas_Bayes 2:0 5:0 S S Grup… Grup…
## 2 2 Thomas_Bayes 1:3 0:1 N N Grup… Grup…
## 3 2 Thomas_Bayes 0:0 0:1 U N Grup… Grup…
## 4 2 Thomas_Bayes 1:2 3:3 N U Grup… Grup…
## 5 3 Thomas_Bayes 2:1 2:1 S S Grup… Grup…
## 6 3 Thomas_Bayes 3:0 1:1 S U Grup… Grup…
## 7 3 Thomas_Bayes 0:2 0:1 N N Grup… Grup…
## 8 3 Thomas_Bayes 4:0 2:0 S S Grup… Grup…
## 9 4 Thomas_Bayes 2:1 0:1 S N Grup… Grup…
## 10 4 Thomas_Bayes 3:0 0:1 S N Grup… Grup…
## # … with 374 more rowsFür Regressionsanalysen brauchen wir hingegen Dummy Variablen. Diese
können wir auf genau dieselbe Art und Weise erstellen. Wenn die
Phase gleich der Gruppenphase ist, schreibe
eine 1, ansonsten eine 0. Das gleiche Prinzip gilt für die Dummy
Variable von KO.
## # A tibble: 384 × 10
## Spieltag Person Tipp Ergebnis Tipp_…¹ Wahre…² Art Phase Gruppe KO
## <dbl> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
## 1 1 Thomas_Bayes 2:0 5:0 S S Grup… Grup… 1 0
## 2 2 Thomas_Bayes 1:3 0:1 N N Grup… Grup… 1 0
## 3 2 Thomas_Bayes 0:0 0:1 U N Grup… Grup… 1 0
## 4 2 Thomas_Bayes 1:2 3:3 N U Grup… Grup… 1 0
## 5 3 Thomas_Bayes 2:1 2:1 S S Grup… Grup… 1 0
## 6 3 Thomas_Bayes 3:0 1:1 S U Grup… Grup… 1 0
## 7 3 Thomas_Bayes 0:2 0:1 N N Grup… Grup… 1 0
## 8 3 Thomas_Bayes 4:0 2:0 S S Grup… Grup… 1 0
## 9 4 Thomas_Bayes 2:1 0:1 S N Grup… Grup… 1 0
## 10 4 Thomas_Bayes 3:0 0:1 S N Grup… Grup… 1 0
## # … with 374 more rows, and abbreviated variable names ¹Tipp_Richtung,
## # ²Wahre_RichtungEine if_else Verzweigung kann immer nur 2 Fälle
abgedecken. Wenn die Bedingung wahr oder eben falsch ist. Was, wenn es
mehr Fälle gibt? Angenommen, wir möchten eine Spalte mit der Punktezahl
pro Spiel erstellen. Bepunkten wollen wir es in traditioneller Manier
mit 3 Punkten für einen komplett richtigen Tipp, 1 Punkt für die
richtige Richtung (Sieg/Niederlage/Unentschieden) und keine Punkte, wenn
weder noch erraten wurde. Das sind drei Fälle. Deswegen benötigen wir
nun case_when(). Hier können beliebig viele Bedingungen
abgefragt werden. Dabei steht die logische Bedingung wieder an erster
Stelle. Was bei der Wahrheit der Bedingung geschehen soll, steht hinter
der Tilde ~. Wenn der Tipp gleich dem Ergebnis ist, gibt es
drei Punkte usw. Die letzte Zeile des Befehls sieht etwas komisch aus.
Dabei funktioniert das TRUE wie das else im
if_else Befehl. Es deckt alle sonstigen
Fälle ab.
tipp_wm %>%
mutate(Punkte = case_when(Tipp == Ergebnis ~ 3,
Tipp_Richtung == Wahre_Richtung ~ 1,
TRUE ~ 0))## # A tibble: 384 × 9
## Spieltag Person Tipp Ergebnis Tipp_Richtung Wahre…¹ Art Phase Punkte
## <dbl> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <dbl>
## 1 1 Thomas_Bayes 2:0 5:0 S S Grup… Grup… 1
## 2 2 Thomas_Bayes 1:3 0:1 N N Grup… Grup… 1
## 3 2 Thomas_Bayes 0:0 0:1 U N Grup… Grup… 0
## 4 2 Thomas_Bayes 1:2 3:3 N U Grup… Grup… 0
## 5 3 Thomas_Bayes 2:1 2:1 S S Grup… Grup… 3
## 6 3 Thomas_Bayes 3:0 1:1 S U Grup… Grup… 0
## 7 3 Thomas_Bayes 0:2 0:1 N N Grup… Grup… 1
## 8 3 Thomas_Bayes 4:0 2:0 S S Grup… Grup… 1
## 9 4 Thomas_Bayes 2:1 0:1 S N Grup… Grup… 0
## 10 4 Thomas_Bayes 3:0 0:1 S N Grup… Grup… 0
## # … with 374 more rows, and abbreviated variable name ¹Wahre_RichtungEs ist Dir sicher aufgefallen, dass wir bisher noch nie den bereits
erwähnten Zuweisungspfeil <- verwendet haben. Das heißt,
dass unsere Rechnungen in Schall und Rauch aus Einsen und Nullen
verschwunden sind. Möchten wir die Ergebnisse der Pipe speichern, müsse
wir die Ergebnisse einer Variable zuordnen. Wenn wir lediglich Spalten
dran gehängt haben, macht es durchaus Sinn, dabei den selben
Datensatznamen zu verwenden. Ansonsten ist ein anderer, aussagekräftiger
Name zu empfehlen wie tipp_wm_bayes falls zum Beispiel nach
allen Tipps von Thomas Bayes gefiltert wurde.
tipp_wm <- tipp_wm %>%
mutate(Punkte = case_when(Tipp == Ergebnis ~ 3,
Tipp_Richtung == Wahre_Richtung ~ 1,
TRUE ~ 0))Wollen wir Spalten logarithmieren oder standardisieren, können wir
dies mit log() respektive scale() machen. Zur
Illustration behalten wir nur die Spalten der Spieltage.
tipp_wm %>%
mutate(Spieltag_lg = log(Spieltag),
Spieltag_z = scale(Spieltag)) %>%
select(Spieltag, Spieltag_lg, Spieltag_z)## # A tibble: 384 × 3
## Spieltag Spieltag_lg Spieltag_z[,1]
## <dbl> <dbl> <dbl>
## 1 1 0 -1.68
## 2 2 0.693 -1.52
## 3 2 0.693 -1.52
## 4 2 0.693 -1.52
## 5 3 1.10 -1.35
## 6 3 1.10 -1.35
## 7 3 1.10 -1.35
## 8 3 1.10 -1.35
## 9 4 1.39 -1.19
## 10 4 1.39 -1.19
## # … with 374 more rowsMit mutate_at() erhält man die Möglichkeit eine Funktion
wie log() gleich auf mehrere Spalten anzuwenden. Dabei
müssen die Spalten im vars() Argument verpackt werden.
## # A tibble: 384 × 9
## Spieltag Person Tipp Ergebnis Tipp_Richt…¹ Wahre…² Art Phase Punkte
## <dbl> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <dbl>
## 1 0 Thomas_Bayes 2:0 5:0 S S Grup… Grup… 0
## 2 0.693 Thomas_Bayes 1:3 0:1 N N Grup… Grup… 0
## 3 0.693 Thomas_Bayes 0:0 0:1 U N Grup… Grup… -Inf
## 4 0.693 Thomas_Bayes 1:2 3:3 N U Grup… Grup… -Inf
## 5 1.10 Thomas_Bayes 2:1 2:1 S S Grup… Grup… 1.10
## 6 1.10 Thomas_Bayes 3:0 1:1 S U Grup… Grup… -Inf
## 7 1.10 Thomas_Bayes 0:2 0:1 N N Grup… Grup… 0
## 8 1.10 Thomas_Bayes 4:0 2:0 S S Grup… Grup… 0
## 9 1.39 Thomas_Bayes 2:1 0:1 S N Grup… Grup… -Inf
## 10 1.39 Thomas_Bayes 3:0 0:1 S N Grup… Grup… -Inf
## # … with 374 more rows, and abbreviated variable names ¹Tipp_Richtung,
## # ²Wahre_RichtungEin paar wichtige Konzepte fehlen uns noch. Dafür schauen wir uns
einen neuen Datensatz namens big_five an. Der Datensatz
beinhaltet soziodemographische Daten wie Alter,
Geschlecht und Herkunft sowie Spalten für die
durchschnittliche Ausprägung von Extraversion,
Neurotizismus, Verträglichkeit und
Gewissenhaftigkeit. Für den fünften Persönlichkeitsfaktor
Offenheit wurde noch kein durchschnittlicher Wert
berechnet. Es gibt 10 Items O1 bis O10, die
die Dimension der Offenheit abgefragt haben.
big_five## # A tibble: 200 × 17
## Alter Gesch…¹ Herku…² Extra…³ Neuro…⁴ Vertr…⁵ Gewis…⁶ O1 O2 O3 O4
## <dbl> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 36 m DE 3 1.9 3.4 3.3 5 1 5 1
## 2 30 f US 3.1 3.4 3.1 2.7 5 3 5 3
## 3 23 m US 3.4 2.4 3.6 3 3 3 5 3
## 4 54 m US 3.3 4.2 3.6 3 2 5 3 4
## 5 24 f US 3 2.8 3 3.3 5 1 5 1
## 6 14 f US 2.8 3.5 3.2 3.3 5 1 5 2
## 7 32 m HK 3.5 3.1 4.2 3 3 1 5 1
## 8 20 m IN 3.5 2.6 3.5 2.7 2 1 3 1
## 9 29 f IN 3 3.7 3.2 2.9 4 1 5 1
## 10 17 m US 3.1 3.6 3 2.9 4 3 4 5
## # … with 190 more rows, 6 more variables: O5 <dbl>, O6 <dbl>, O7 <dbl>,
## # O8 <dbl>, O9 <dbl>, O10 <dbl>, and abbreviated variable names ¹Geschlecht,
## # ²Herkunft, ³Extraversion, ⁴Neurotizismus, ⁵Vertraeglichkeit,
## # ⁶GewissenhaftigkeitSomit stoßen wir auch schon auf eine sehr häufige Problemstellung.
Das Berechnen von Mittelwerten oder Summen über mehrer Spalten pro
Person (also zeilenweise). Dafür wählen wir uns zuerst die
interessierenden Spalten wie gewohnt mit select() aus. Wenn
die Spalten nebeneinander liegen, kann man auch einfach einen
Doppelpunkt benutzen.
Der Doppelpunkt liest sich hier zum Beispiel als O1 bis O10. Erst
einmal ausgewählt, ist der Rest schnell erledigt. Wir erstellen mit
mutate() eine neue Spalte namens Offenheit und
erhalten mit rowMeans() die Mittelwerte pro Zeile (row),
also pro Person. Es gibt eine äquivalente Funktion
rowSums() zum Summieren. Anschließend möchten wir die
einzelnen Items nicht im Datensatz, sondern nur die soziodemographischen
Daten und die fünf Dimensionen der Persönlichkeitsfaktoren behalten.
## # A tibble: 200 × 8
## Alter Geschlecht Herkunft Extraversion Neurotizismus Vertra…¹ Gewis…² Offen…³
## <dbl> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 36 m DE 3 1.9 3.4 3.3 3.6
## 2 30 f US 3.1 3.4 3.1 2.7 3.8
## 3 23 m US 3.4 2.4 3.6 3 3.2
## 4 54 m US 3.3 4.2 3.6 3 2.9
## 5 24 f US 3 2.8 3 3.3 3.5
## 6 14 f US 2.8 3.5 3.2 3.3 3.6
## 7 32 m HK 3.5 3.1 4.2 3 2.7
## 8 20 m IN 3.5 2.6 3.5 2.7 2.7
## 9 29 f IN 3 3.7 3.2 2.9 3.6
## 10 17 m US 3.1 3.6 3 2.9 3.5
## # … with 190 more rows, and abbreviated variable names ¹Vertraeglichkeit,
## # ²Gewissenhaftigkeit, ³OffenheitDie Reihenfolge der in select() ausgewählten Spalten
entscheidet übrigens über die Reihenfolge der Spalten. Alternativ zum
Auswählen der Spalten, die man behalten möchte, kann man auch einfach
die ungewünschten Spalten O1 bis O10
entfernen.
Beachte die Klammern um O1:O10. Was ist eigentlich, wenn
man nur Spalten mit Zahlen im Datensatz behalten möchte? Mit
select_if() ist eine speziell auf solche Aufgaben
zugeschnittene Funktion verfügbar. Wenn die Spalte
numerisch ist (is.numeric), wird sie behalten.
## # A tibble: 200 × 15
## Alter Extravers…¹ Neuro…² Vertr…³ Gewis…⁴ O1 O2 O3 O4 O5 O6
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 36 3 1.9 3.4 3.3 5 1 5 1 4 1
## 2 30 3.1 3.4 3.1 2.7 5 3 5 3 4 1
## 3 23 3.4 2.4 3.6 3 3 3 5 3 4 1
## 4 54 3.3 4.2 3.6 3 2 5 3 4 3 3
## 5 24 3 2.8 3 3.3 5 1 5 1 5 1
## 6 14 2.8 3.5 3.2 3.3 5 1 5 2 2 1
## 7 32 3.5 3.1 4.2 3 3 1 5 1 5 1
## 8 20 3.5 2.6 3.5 2.7 2 1 3 1 4 1
## 9 29 3 3.7 3.2 2.9 4 1 5 1 5 1
## 10 17 3.1 3.6 3 2.9 4 3 4 5 4 2
## # … with 190 more rows, 4 more variables: O7 <dbl>, O8 <dbl>, O9 <dbl>,
## # O10 <dbl>, and abbreviated variable names ¹Extraversion, ²Neurotizismus,
## # ³Vertraeglichkeit, ⁴GewissenhaftigkeitEin weiterer praktischer Sonderfall ist select_at(). Die
Funktionsweise ist die selbe wie bei mutate_at(). Der
Vorteil ist die Integration von starts_with() und
ends_with() womit man, wie der Name bereits suggeriert,
Anfangs- respektive Endbuchstaben oder ganze Wörter auswählen kann. Es
werden also nur die Spalten behalten, die mit der Zeichenkombination
starten oder enden. Hier erhalten wir alle Spalten mit dem
Anfangsbuchstaben E. Also hier nur Extraversion.
big_five %>%
select_at(vars(starts_with("E")))## # A tibble: 200 × 1
## Extraversion
## <dbl>
## 1 3
## 2 3.1
## 3 3.4
## 4 3.3
## 5 3
## 6 2.8
## 7 3.5
## 8 3.5
## 9 3
## 10 3.1
## # … with 190 more rowsBreites vs. langes Datenformat
Das lange Datenformat ist eins der wichtigsten Konzepte. Man benötigt es für jeden Graphen und für manche induktiven Verfahren. Man erhält im Großteil der Fälle beim Erheben Daten im breiten Format. Um das besser zu veranschaulichen schauen wir uns einmal an, wie unsere Tipps zur WM in breitem Format aussehen würden.
tipp_wm_wide## # A tibble: 64 × 7
## Thomas_Bayes Egon_Pearson Jerzy_Neyman Alice Bob Charlie Ergebnis
## <chr> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 2:0 1:0 2:1 2:1 3:1 2:1 5:0
## 2 1:3 0:0 0:2 1:2 0:2 0:2 0:1
## 3 0:0 0:2 1:1 1:1 2:1 1:1 0:1
## 4 1:2 1:1 2:1 2:4 2:2 1:0 3:3
## 5 2:1 NA 2:0 2:1 2:0 2:1 2:1
## 6 3:0 NA 3:1 2:1 2:1 4:0 1:1
## 7 0:2 1:2 0:2 1:2 2:1 1:2 0:1
## 8 4:0 1:2 3:0 1:0 3:0 4:0 2:0
## 9 2:1 0:0 1:1 1:1 0:1 1:0 0:1
## 10 3:0 1:0 2:0 3:1 3:1 2:0 0:1
## # … with 54 more rowsIn jeder Spalte sind hier die Tipps der jeweiligen Person. Was bedeutet es nun, diesen Datensatz lang zu machen?
## # A tibble: 384 × 3
## Ergebnis Person Tipp
## <chr> <chr> <chr>
## 1 5:0 Thomas_Bayes 2:0
## 2 0:1 Thomas_Bayes 1:3
## 3 0:1 Thomas_Bayes 0:0
## 4 3:3 Thomas_Bayes 1:2
## 5 2:1 Thomas_Bayes 2:1
## 6 1:1 Thomas_Bayes 3:0
## 7 0:1 Thomas_Bayes 0:2
## 8 2:0 Thomas_Bayes 4:0
## 9 0:1 Thomas_Bayes 2:1
## 10 0:1 Thomas_Bayes 3:0
## # … with 374 more rowsWie man sieht, werden nun alle Ergebnisse, Personen und Tipps jeweils
in eine Spalte geschrieben. Wir erreichen dies mit
gather(). Das erste Arugment ist der Name der neuen
Spalten, die die Werte der Spaltennamen enthält. Also hier die Personen.
Das zweite Argument beschreibt die Werte innerhalb der Spalten, die wir
hier als Tipp bezeichnen. Das dritte Argument sind nun die
Spalten, die zusammen gesammelt werden sollen. Wir wollen alle Personen
– also von Thomas Bayes bis Charlie – in der Spalte Person
haben. Weshalb wir das brauchen, wird in den folgenden Kapiteln noch
klarer. Das Prinzip hinter gather() ist etwas abstrakt und
verlangt am Anfang erst einmal Gewöhnung. Wenn man sich nicht sicher
ist, welcher Name nun wozu gehört kann man gut und gerne auch erst
einmal sowas wie
## # A tibble: 384 × 3
## Ergebnis test test1
## <chr> <chr> <chr>
## 1 5:0 Thomas_Bayes 2:0
## 2 0:1 Thomas_Bayes 1:3
## 3 0:1 Thomas_Bayes 0:0
## 4 3:3 Thomas_Bayes 1:2
## 5 2:1 Thomas_Bayes 2:1
## 6 1:1 Thomas_Bayes 3:0
## 7 0:1 Thomas_Bayes 0:2
## 8 2:0 Thomas_Bayes 4:0
## 9 0:1 Thomas_Bayes 2:1
## 10 0:1 Thomas_Bayes 3:0
## # … with 374 more rowsschreiben und test und test1 anschließend
in passende Spaltennamen ändern. Das Gegenstück zu gather()
ist spread(), allerdings ist die Funktion kein perfekter
Gegenspieler. Häufiger treten Fehler auf. Gut für uns, dass es praktisch
nicht vorkommt, lange Formate breit machen zu wollen.
Den größten Teil hast du nun bereits hinter Dir. Bevor Du dich jedoch euphorisch auf die Übungsaufgaben stürzst, noch ein paare Worte zu Fehler- und Warnmeldungen.
Fehler- und Warnmeldungen
Es gibt einen großen Unterschied zwischen Fehler- und Warnmeldungen. Wie der Name bereits suggeriert, stoppen Fehlermeldungen den Code, während Warnmeldungen ein Ergebnis zurückgeben und nur auf eine Ungereimtheit hinweisen. Es ist also sehr wichtig, die roten Meldungen in der Konsole genau zu lesen. Wichtig ist, beim roten Text nicht gleich in Panik zu geraten, sondern entspannt die Meldung durchzulesen und falls man nicht weiter weiß, die Meldung zu googeln.
Übungen
Nun geht’s an die Übung. Kopiere den R Befehl in Deine Konsole und es startet sich ein interaktives Tutorial in Deinem Standardbrowser.
learnr::run_tutorial("wiederholung", package = "rBasics")Der interaktive Übungssatz beinhaltet verschiedene Übungen, die das
Verständnis des Gelernten prüfen sollen. Dabei kann man sich Hinweise
holen, wenn man nicht weiterkommt. Bei Multiple Choice Fragen wird
sofort geprüft, ob die Antwort korrekt ist. Um zu erfahren, ob der
geschrieben Code richtig ist, kann man mehrmals auf Hints
drücken, da der letzte Hinweis immer die Lösung beinhaltet. Der
Fortschritt wird automatisch auf Deinem Computer gespeichert, sodass Du
zu einem späteren Zeitpunkt die Übungen fortführen kannst. Viel Erfolg
und vor allem viel Spaß!